揭秘“超级细菌”进化机制 ——北京大学人民医院检验科王辉教授团队最新成果在国际权威期刊发表
近日,由北京大学人民医院王辉教授团队在Drug Resistance Updates杂志(IF="24.3)上在线发表了题为Expansion" and transmission dynamics of high risk carbapenem-resistant Klebsiella pneumoniae subclones in China: An epidemiological,spatial,genomic analysis(“中国碳青霉烯耐药肺炎克雷伯菌高危亚克隆的扩张和传播动力:空间流行病学、基因组分析”)的研究成果。

碳青霉烯耐药的肠杆菌目细菌被认为是临床上的“超级细菌”。一旦感染,治疗非常困难,多药耐药且病死率高。在我国,碳青霉烯耐药肺炎克雷伯菌(CRKP)是“超级细菌”的主要菌种。类似于人类的姓氏、细菌也有自己的“家谱”,我国所流行的CRKP祖先是哪里的?哪些因素驱动了它的传播和进化还不得而知。
研究团队通过大规模纵向研究,揭示了“超级细菌”——CRKP的主要种群ST11最早可能来自于美洲大陆,2010年前后传入我国,目前已进化成为一个高危亚克隆——“BMPPS”突变株,并探究了其在临床的高度扩张性及其进化的驱动因素。
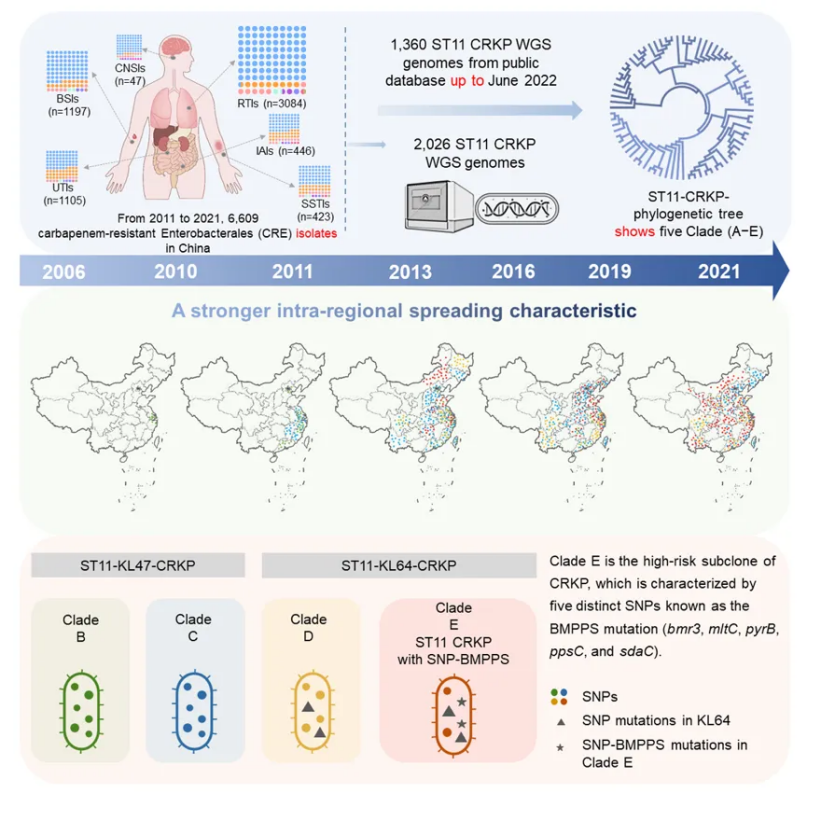
该研究属于中国碳青霉烯耐药肠杆菌目监测网(CRE Network)的一部分。总共收集了,2011年-2021年我国CRE-Network项目中28个省市96家医院的6609碳青霉烯耐药肠杆菌目细菌,其中4718株CRKP,ST11 CRKP占比75%。
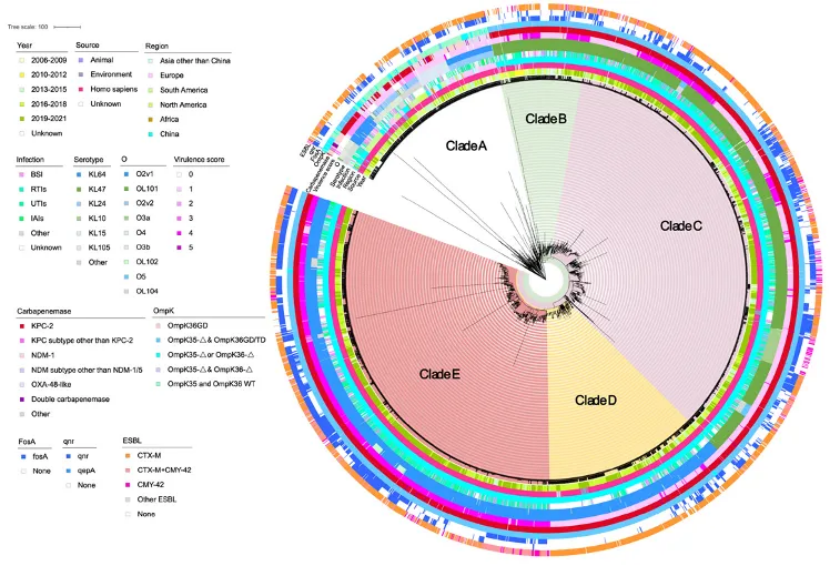
传统细菌“姓氏”的追踪溯源常使用多位点序列分析(MLST),一般通过7个管家基因进行比对,而全基因组测序(WGS)技术可大大的提高菌株溯源的分辨率。研究团队整合了CRE-Network中2026株ST11 CRKP基因组以及公共数据库中1360株ST11 CRKP的基因组,绘制了全球范围内最大的ST11 CRKP的系统发育树(图1)。ST11 CRKP在我国以两个血清型(KL47和KL64)为主的进化分支,其中KL64血清学逐渐替代KL47成为主要血清型。KL64分支还可再细分为2个分支(Clade D和Clade E),99%以上Clade E菌株携带毒力及重要耐药基因。时间纵向来看,Clade E在CRKP种群中正在逐渐取代其他亚克隆成为主要流行克隆,且存在较大规模的区域性传播事件。
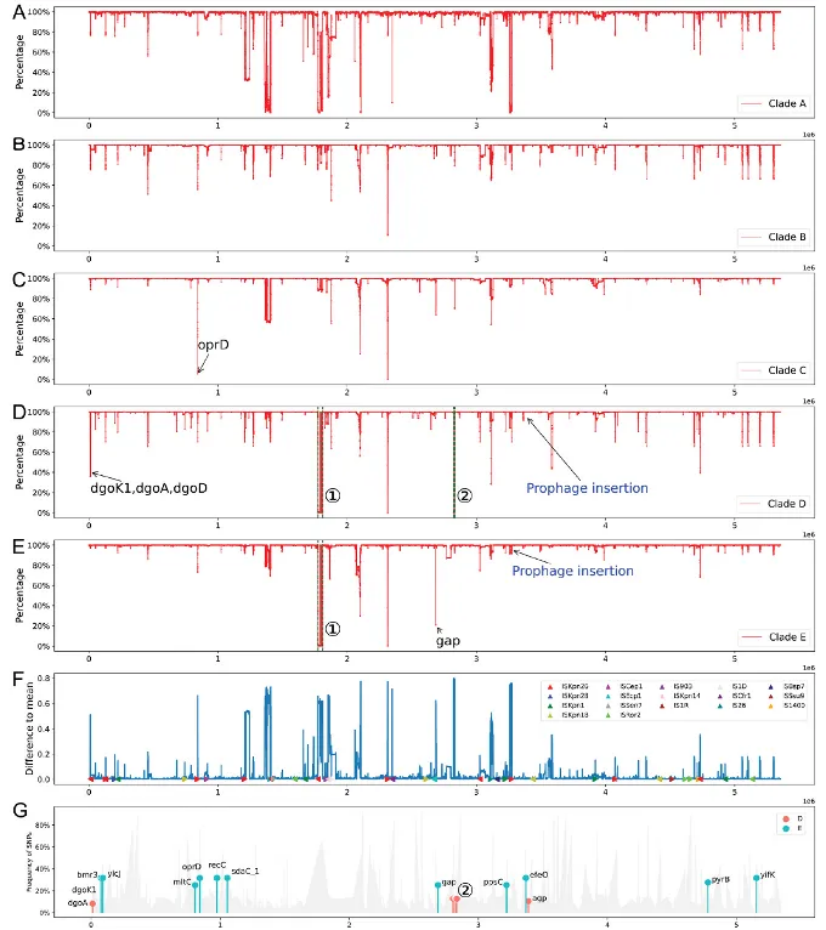
通过比较基因组学分析(图2),不同于其他Clade,Clade E存在5个独特的基因位点变异(SNV)。因此,以“BMPPS”—— (bmr3, mltC, pyrB, ppsC, sdaC)对该高危克隆进行命名(谐音“Bombs”),研究团队利用CRISPR-Cas9基因编辑技术成功构建了pyrB,ppsC及mltC三个基因的敲除株和突变株,结果显示,突变可明显提高非Clade E的亚克隆菌株对抗巨噬细胞吞噬能力及补体C3结合力。研究还调查了445名CRKP血流感染患者的生存分析,≥65岁患者“BMPPS”菌株血流感染病死率高达54.5%,且相对于其他亚克隆“BMPPS”菌株具有较强的抗巨噬细胞吞噬能力及竞争生长能力。
综上,该研究对我国CRKP的重要流行亚克隆进行细分,利用比较基因组学、分子克隆试验证明了我国CRKP流行的“BMPPS”突变株的高度扩张性,且具有严重的临床危害性。同时,也证明了该高危亚克隆的关键突变位点在其进化、演化中的作用,BMPPS也可成为未来诊断和治疗的重要靶点。
该研究并列第一作者为来自北京大学人民医院检验科王启副主任技师、王若冰副研究员、博士生王舒意和博士生张安汝,北京大学人民医院检验科王辉教授为文章的通讯作者。
(人民医院)
编辑:玉洁


